-
Takara Cut-Site Navigatorを開きます。
-
解析を行う配列の名称を、「Sample Name」欄に入力してください。(必須ではありません。)

入力した名称は、解析結果を出力する際のFile名に使用されます。
-
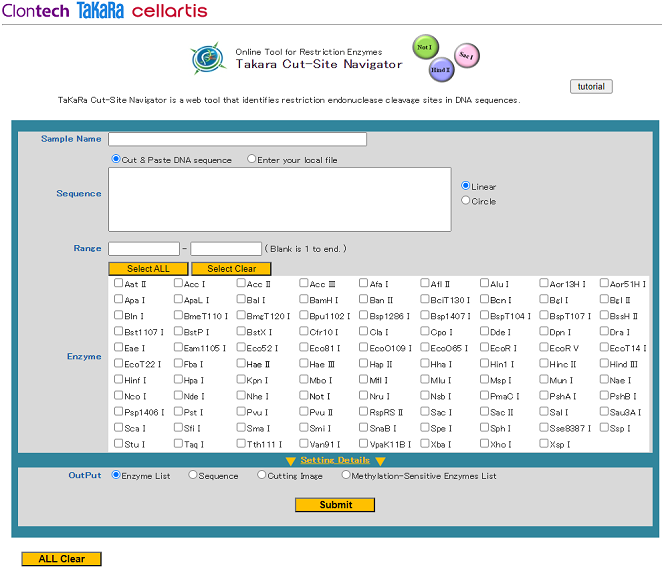
制限酵素検索を行う配列情報を、「Sequence」欄に入力します。
入力は、以下に示す「Cut & Paste DNA Sequence」または「Enter your local file」で行います。どちらかを選択して、配列情報を入力してください。
なお、入力する配列情報がPlasmidなどの環状配列の場合「Circle」を、Fragment状(直線状)の場合「Linear」を選択してください。
NやRなどの核酸コードが利用できます。
*「Cut & Paste DNA Sequence」:配列情報を直接入力する方法
配列情報をSequence入力欄にCopy&Pasteしてください。
*「Enter your local file」:配列情報Fileを選択して入力する方法
右側のボタンをクリックすると、File選択のためのWindowが表示されます。
該当する配列情報FileをLocal Discから選択してください。
使用可能なファイル形式は、テキスト形式、fasta形式、GenBank形式です。
-
検索条件の設定
「Takara Cut-Site Navigator」では制限酵素を選択する際に、いろいろな条件で絞り込むことが可能です。
絞り込み条件としては、以下の6種類があります。
a) Range
b) Enzyme
c) Cutting Type
d) Search Methylation-Sensitive Enzymes
e) Recognition Sequence Length
f) Frequency
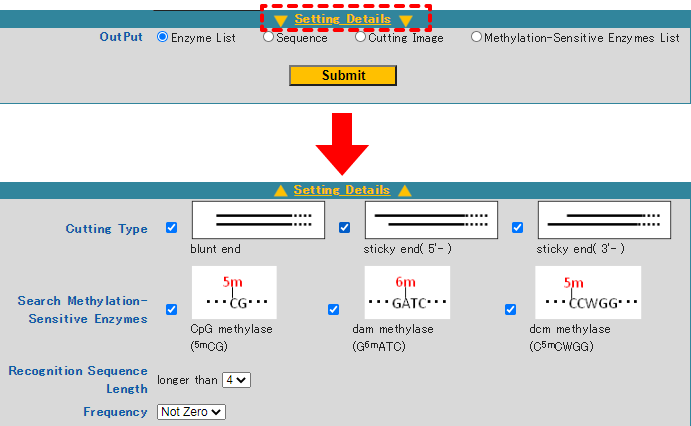
c)~f)を設定するためにはSetting Detailes をクリックします。
各条件の詳細に関しては、以下を参照してください。
a) Range

入力した配列情報に対して、実際の制限酵素検索に使用する範囲を指定することができます。
指定する範囲を、配列Positionの数値で入力してください。
b) Enzyme
候補となる制限酵素が限定される場合、その特定の制限酵素で検索することが可能です。
検索したい制限酵素の左側のCheck欄にチェックを入れてください。
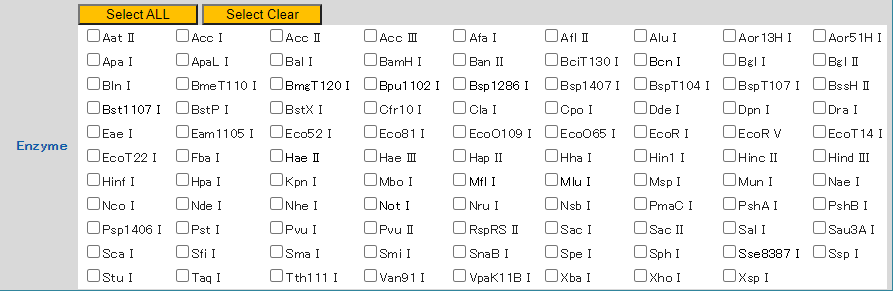
ちょっと便利な機能 !!
制限酵素名にカーソルを合わせると、制限酵素切断サイトの情報が表示されます。
制限酵素が認識する切断サイトを確認したい場合に便利な機能です。
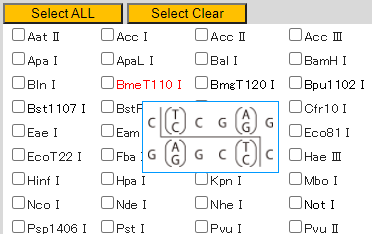
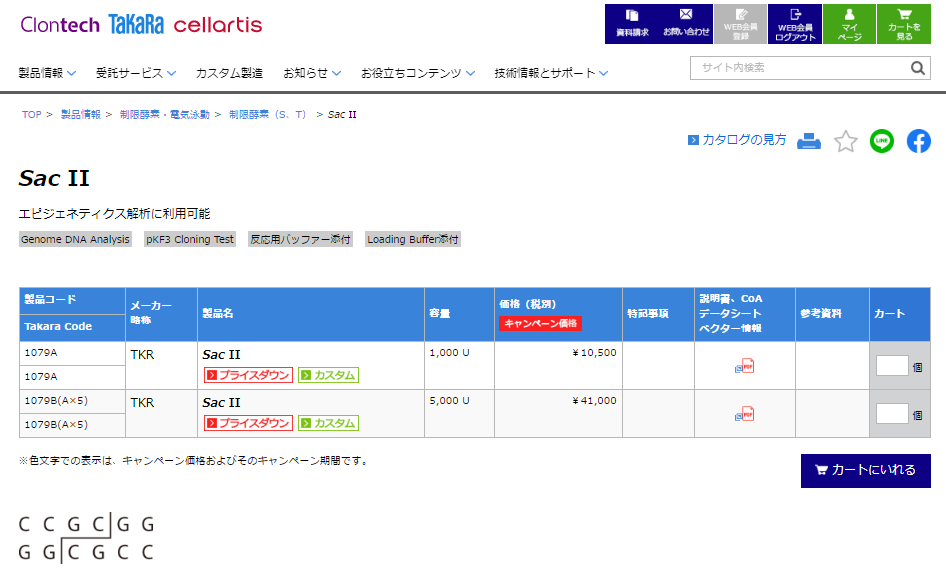
制限酵素名をクリックすると、該当するTakara制限酵素製品のウェブカタログページを開くことができます。
注目している制限酵素の詳細情報を確認する際に便利です。
c) Cutting Type
制限酵素で切断した際の切断形状を検索条件として設定することが出来ます。
Blunt end : 平滑末端、Sticky end(5'-) : 5'突出末端、Sticky end(3'-) : 3'突出末端の3種類から複数選択が可能です。

なお、デフォルト設定は3種類すべてを選択した状態です。
d) Search Methylation-Sensitive Enzymes
制限酵素の中には、そのDNA認識配列中の塩基がメチル化を受けると、メチル化された塩基の種類および位置によってDNAを切断できなくなるものがあります。
この検索ツールでは、こういったメチル化感受性制限酵素であるかどうかを、検索条件として設定することができます。

検索条件として、設定できるメチル化酵素は以下の3種類です。
CpG methylase(5mCG) : CpG methylase(5mCG)によるメチル化の影響をうける制限酵素(哺乳類ゲノムのDNAメチル化解析)
dam methylase(G6mATC) : dam methylase (G6mATC) によるメチル化の影響を受ける制限酵素(大腸菌由来のmethylase)
dcm methylase(C5mCWGG) : dcm methylase (C5mCWGG) によるメチル化の影響を受ける制限酵素(大腸菌由来のmethylase)
各チェック欄にチェックを入れることにより、塩基配列切断活性に影響がある制限酵素情報が、解析結果に表示されます。
「Enzyme List」で結果を表示した場合、下記の赤囲みの部分にメチル化感受性制限酵素の情報が表示されます。
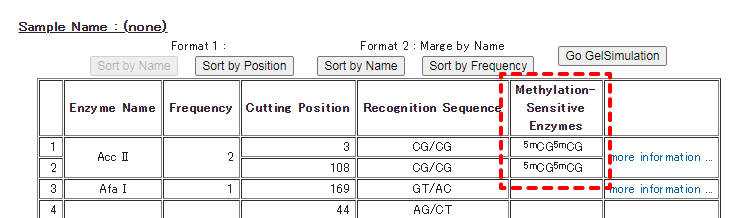
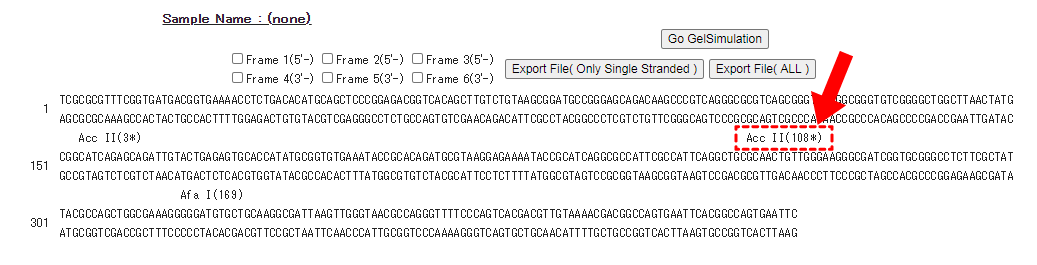
「Sequence」で結果を表示した場合、メチル化感受性制限酵素は*で表示されます。
「Methylation-Sensitive Enzymes List」で結果を表示した場合、メチル化感受性制限酵素のみを選択して表示されます。
e) Recognition Sequence Length
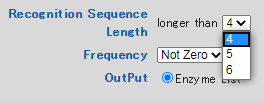
制限酵素が認識する配列の長さで、検索条件を設定することができます。
ドロップダウンメニューを開いて、「4」「5」「6」の3種類から選択します。
4 : 4 bp以上の配列を認識する制限酵素を選択
5 : 5 bp以上の配列を認識する制限酵素を選択
6 : 6 bp以上の配列を認識する制限酵素を選択
f) Frequency
制限酵素が該当配列を切断する際の切断頻度で、検索条件を設定することができます。
ドロップダウンメニューを開いて、「Not Zero」「0~5」の7種類から選択します。
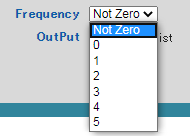
Not Zero : 切断頻度を検索条件に設定しない場合に使用します。
0 : 対象配列中に切断箇所が存在しない制限酵素を検索します。
1 : 対象配列中に切断箇所が1箇所存在する制限酵素を検索します。
2 : 対象配列中に切断箇所が2箇所存在する制限酵素を検索します。
3 : 対象配列中に切断箇所が3箇所存在する制限酵素を検索します。
4 : 対象配列中に切断箇所が4箇所存在する制限酵素を検索します。
5 : 対象配列中に切断箇所が5箇所存在する制限酵素を検索します。
-
検索条件の設定が完了したら、「OutPut」で検索表示方法を選択します。
表示形式は、「Enzyme List」「Sequence」「Cutting Image」「Methylation-Sensitive Enzymes List」の4つから選択できます。

結果はNやRなどの核酸コードを反映しているものです。取扱いには十分ご注意ください。
各表示方法の詳細は、以下を参照してください。
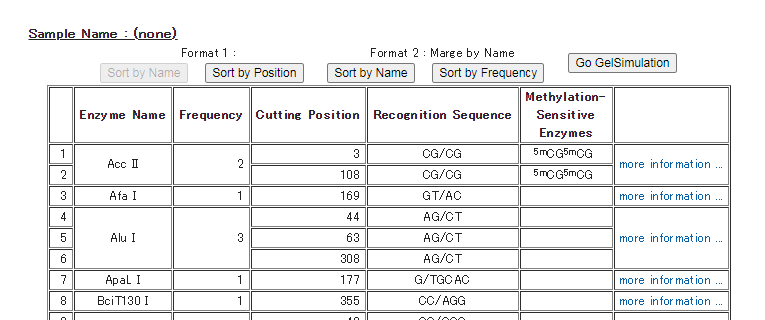
*「Enzyme List」
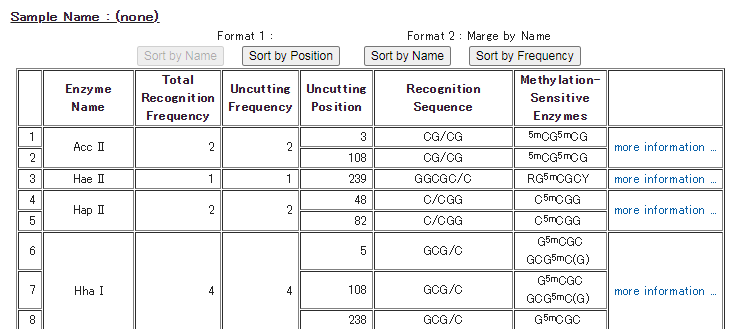
検索条件に該当した制限酵素をリスト形式で表示します。
Listに表示された項目は、制限酵素名や切断頻度でSortすることも可能です。
(「Sort by Name」「Sort by Frequency」のボタンをクリックしてください。)
*「Sequence」
配列情報を表示し、切断箇所に該当する制限酵素名を表示する形式です。
(制限酵素名の右側の括弧内に、切断位置のPosition番号を表記しています。)
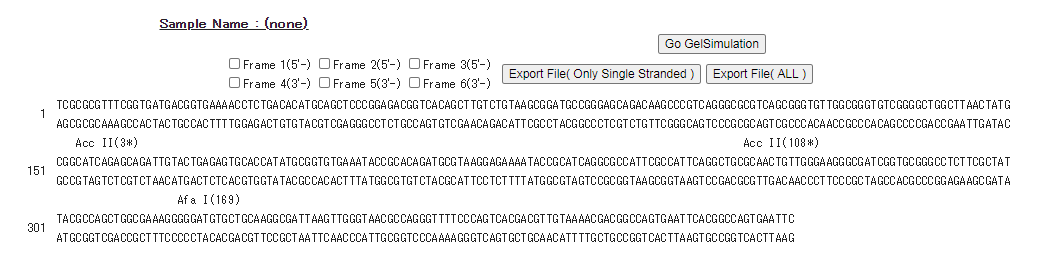
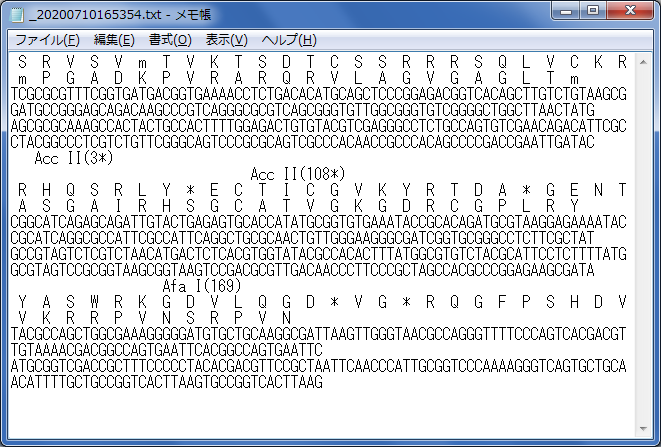
塩基配列情報に対応するアミノ酸配列を表示させたい場合、「Frame 1~6」にチェックを入れることでアミノ酸配列を表示させることができます。

*は終止コドン・予測不可能なアミノ酸を示します。
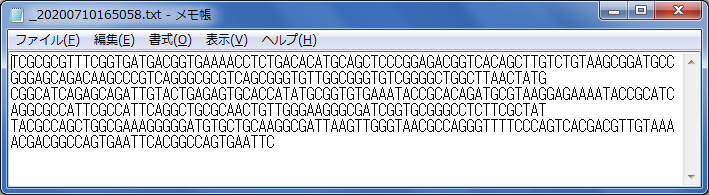
表示された解析結果は、以下のボタンをクリックすることで、Local PCにテキスト形式で出力することができます。

各出力パターンは、以下を参照してください。
「Export File(Only Single Stranded)」:指定された領域のSingle Strandの配列情報のみを出力します。
「Export File(ALL)」:画面に表示されている情報(塩基配列情報、アミノ酸配列情報、制限酵素情報)をすべて出力します。
*「Cutting Image」
制限酵素で切断される場所が、対象配列のどの部分に当たるかを模式的に表示する表示形式です。
Cutting Image中の注目したい部分を2カ所クリックして×を表示した後、「Submit」ボタンをクリックすると、指定された部分の模式図を拡大することができます。
拡大しすぎた場合、「Range Clear」ボタンをクリックし、配列指定範囲(Range)を消去した後、「Submit」ボタンをクリックすれば、拡大開始前の初期の配列範囲に戻すことが可能です。
なお、切断箇所が重なる場合、制限酵素名も重なって表示されることがありますが、不具合ではありません。
適宜 拡大機能を使用して、内容を確認してください。
どうしても、重なった制限酵素名を確認したい場合は、OutPut形式を「Sequence」に切り替えることで確認できます。

*「Methylation-Sensitive Enzymes List」
「Methylation-Sensitive Enzymes」でメチル化感受性制限酵素の検索条件を設定している場合、この検索条件で該当する制限酵素のみを選択して表示します。

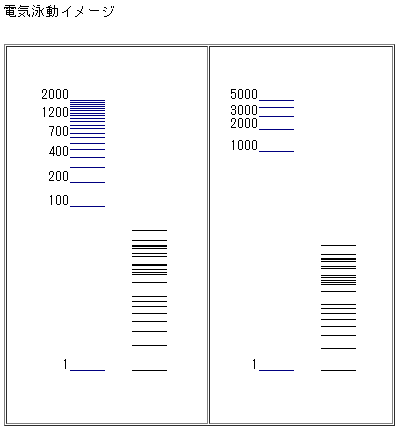
*「Go GelSimulation」
各表示形式の右側に表示される「Go GelSimulation」をクリックすると、選択された制限酵素で対象配列を切断した際の電気泳動バンドイメージを確認することが出来ます。
左端の青色のバンドは、バンドサイズを示すマーカーイメージです。
*「All Clear」
“All Clear”ボタンをクリックすると、検索条件および入力情報をすべて初期設定にリセットすることができます。